RNAワールドの酸性条件下での進化: the RNA world evolution at acidic pH
RNAワールドは、酸性条件下で進化してきた可能性が高い(Bernhardt and Tate 2012)。酸性環境下でのRNA分子の安定性と酸性で機能するリボザイムの発見がそれを支持している。
まず、RNAを構成する主要な分子が酸性条件下での方が安定である。RNAはPH4-5で最も安定であるが、アルカリ条件では不安定である(Javinen et al. 1991)。つまり、PH>6でRNAの燐酸ジエステル結合が加水分解を始めるが、酸性条件においては、PH<2まで加水分解しない(Oivanen et al. )。次に、tRNAとその前駆体と考えられているヘアピンRNAは、中性とアルカリPHで不安定であ(Welfenden 1963; Schnber 1973; De Giulio 1992; Bernhardt 2008)。現生生物では、RNAをリボゾームに運ぶたんぱく質伸長因子Ef-TuがRNAのアミノアシル-RNA結合を保護しているが、それがまだない初期の遺伝情報からのたんぱく質合成においては、これらの無水燐酸結合の安定性は遺伝情報によるたんぱく質合成に本質的に重要と考えられている(Nissen et al 1995)。さらに、ペプチドの結合はPH4-7.5で安定であり、PH=6で最も安定になる(Pascal et al 2005)。
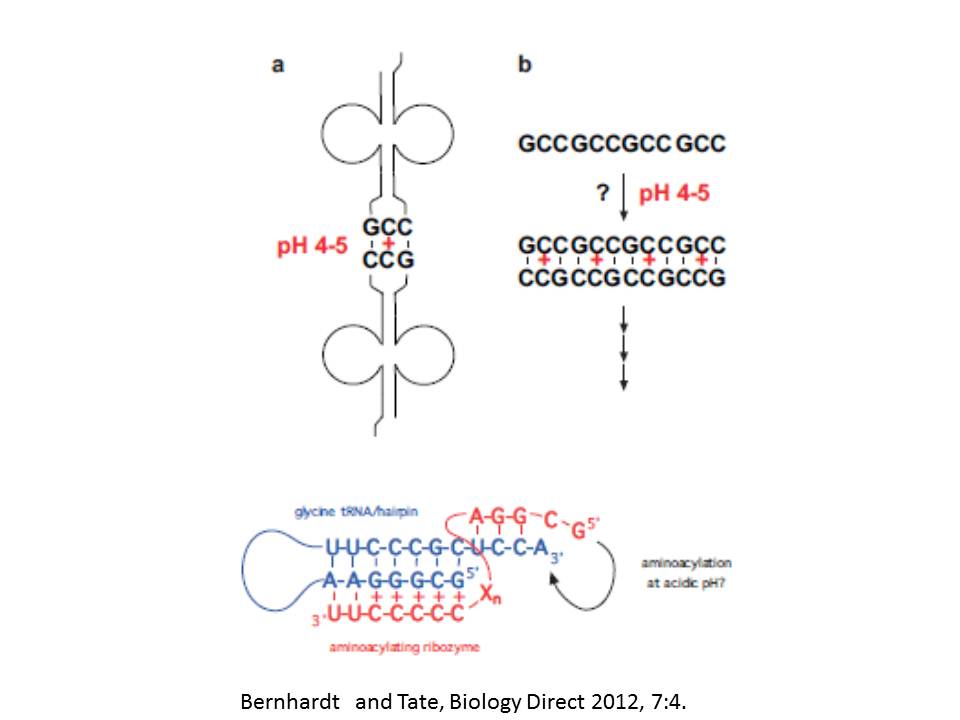
次に、酸性PHで自己複製と自己アミノアシル化の機能を持つリボザイムが試験管内選択により得られた(Miyamoto 2001; 2005; Kuhne et al. 2003; Jayasena 1997; Kumer et al. 2001)。これらは、RNAワールドの維持に必須である。RNAレプリカーゼの出現の前に、短いRNA鎖による非酵素的なRNA複製が始まったはずである。ところが一対の相補的なRNA鎖の一つが、もう一つの鋳型になって、その配列を繰り返し複製する仕組みが分からなかった(Robertson et al 2011)。Cが多いRNA鎖たとえばCCGCCが注目を集めている(Inoue et al. 1984)。しかし、Cに富む配列は中性条件では、Gに富むコピーを作り、より高次な構造ができて複製が停止してしまう。
ところが、酸性条件では、以下に説明するような普通でない塩基対ができるので、それがこの問題を解決できる可能性がある。たとえば、大腸菌のtRNA-Gly(GCC)は、PH4-5の酸性条件のみで、準自己相補的なアンチコドンと結合して、二倍体を作る(Romby et al. 1984)。この時、中央のCが半プロトン化して一つのプロトンを共有して結合している。したがって、GCCの繰り返し配列は、自分自身を複製するよい鋳型になりえる(Durant 1999; Merough 1999; Chirkova 2010; Venditti 2009; Bink 2002)。同様に酸性条件下では、半プロトン化したCとAとの間の結合C-A(+)の可能である。つまり、GCC配列はGAC繰り返し配列もしくは、GCC/GACが混じった配列の鋳型になりえる。
De Giulo (1992)は、tRNAは、その約半分の長さのヘアピンRNA鎖から始まって進化したと考えている。Chumachenko and Novikov, and Yarus (2009)は、24個の塩基を持つ自分でアミノアシル化能力を持つリボザイムを合成した。そのうち3つの塩基のみ保存されていた。このリボザイムを順に切り取ってゆくと、5ヌクレオチド鎖リボザイムが、4ヌクレオチドRNAをアミノアシル化することを見出した。これは、tRNA/ヘアピンのアミノアシルステムの特定認識は、配列操作なしでできることを示唆している。このような認識は、酸性条件下のみで作られるピリミジン-プリン-ピリミジン三重鎖の形成により、可能かもしれない(Holland 1996)。つまり、アミノアシル化しようとしているリボザイムのポリミジン配列が、プリンに富む5末端側の配列の塩基に特有の相互作用を通して、tRNA/ヘアピンのアミノアシル部ステムの中にある運用部位を認識するのである。
実際、最初に進化したtRNAと考えられているtRNA-Gly(GCC)は(Bernhardt et al. 2008)、GCGGGAAというプリンに富む配列を持っている。他の多くのtRNAも同様である。アミノアシルステムの中のプリンに富む違った配列は、それに相補的なピリミジン配列によって認識される。その先には対応するアミノ酸が結合される。
Jarvinen P, Oivanen M, Lonnberg H: Interconversion and phosphoester hydrolysis of 2’,5’- and 3’,5’-dinucleoside monophosphates: kinetics and mechanisms. J Org Chem 1991, 56:5396-5401.
Bernhardt, H.S. and Tate, W.P., Primordial soup or vinaigrette: did the RNA world evolve at acidic pH?, Biology Direct 2012, 7:4.
Oivanen M, Kuusela S, Lonnberg H: Kinetics and mechanisms for the cleavage and isomerization of the phosphodiester bonds of RNA by Bronsted acids and bases. Chem Rev 1998, 98:961-990.
Miyamoto Y, Teramoto N, Imanishi Y, Ito Y: In vitro evolution and characterization of a ligase ribozyme adapted to acidic conditions: effect of further rounds of evolution. Biotechnol Bioeng 2005, 90:36-45.
Miyamoto Y, Teramoto N, Imanishi Y, Ito Y: In vitro adaptation of a ligase ribozyme for activity under a low-pH condition. Biotechnol Bioeng 2001, 75:590-596
Di Giulio M: On the origin of the transfer RNA molecule. J Theor Biol 1992, 159:199-214.
Bernhardt HS, Tate WP: Evidence from glycine transfer RNA of a frozen accident at the dawn of the genetic code. Biol Direct 2008, 3:53.
Wolfenden R: The mechanism of hydrolysis of amino acyl RNA.
Biochemistry 1963, 2:1090-1092.
Schuber F, Pinck M: On the chemical reactivity of aminoacyl-tRNA ester bond. I. Influence of pH and nature of the acyl group on the rate of hydrolysis. Biochimie 1974, 56:383-390.
Nissen P, Kjeldgaard M, Thirup S, Polekhina G, Reshetnikova L, Clark BF, Nyborg J: Crystal structure of the ternary complex of Phe-tRNAPhe, EFTu, and a GTP analog. Science 1995, 270:1464-1472.
Pascal R, Boiteau L, Commeyras A: From the prebiotic synthesis of α-amino acids towards a primitive translation apparatus for the synthesis of peptides. Top Curr Chem 2005, 259:69-122.
Ferris JP, Usher DA: Origins of life. In Biochemistry. Edited by: Zubay G. Reading, MA: Addison-Wesley; 1983:1191-1241.
Robertson MP, Joyce GF: The origins of the RNA world. In RNA Worlds. Edited by: Atkins JF, Gesteland RF, Cech TR. Cold Spring Harbor, NY: Cold Spring Harbour Press; 2010:21-42.
Inoue T, Joyce GF, Grzeskowiak K, Orgel LE, Brown JM, Reese CB: Templatedirected synthesis on the pentanucleotide CpCpGpCpC. J Mol Biol 1984, 178:669-676.
Romby P, Westhof E, Moras D, Giege R, Houssier C, Grosjean H: Studies on anticodon-anticodon interactions: hemi-protonation of cytosines induces self-pairing through the GCC anticodon of E. coli tRNA-Gly. J Biomol Struct Dyn 1986, 4:193-203.
Durant PC, Davis DR: Stabilization of the anticodon stem-loop of tRNALys,3 by an A+-C base-pair and by pseudouridine. J Mol Biol 1999, 285:115-131.
Meroueh M, Chow CS: Thermodynamics of RNA hairpins containing single internal mismatches. Nucleic Acids Res 1999, 27:1118-1125.
Chirkova A, Erlacher MD, Clementi N, Zywicki M, Aigner M, Polacek N: The role of the universally conserved A2450-C2063 base pair in the ribosomal peptidyl transferase center. Nucleic Acids Res 2010, 38:4844-4855.
Venditti V, Clos L, Niccolai N, Butcher SE: Minimum-energy path for a U6 RNA conformational change involving protonation, base-pair rearrangement and base flipping. J Mol Biol 2009, 391:894-905.
Bink HH, Hellendoorn K, van der Meulen J, Pleij CW: Protonation of non-Watson-Crick base pairs and encapsidation of turnip yellow mosaic virus RNA. Proc Natl Acad Sci USA 2002, 99:13465-13470.
Holland JA, Hoffman DW: Structural features and stability of an RNA triple helix in solution. Nucleic Acids Res 1996, 24:2841-2848.
書籍版『科学はひとつ』のご案内
戎崎俊一 著
学而図書/四六判 並製320頁/本体2,400円+税
12年にわたり執筆されてきた記事を精選し、「地震と津波防災」など全9章に再編。すべての章に著者書き下ろしの解説を加えて集成した一冊。